News


| [%article_date_notime_dot%] [%new:New%] |
[%title%] |
|---|---|

Development dev.204239. (2025)
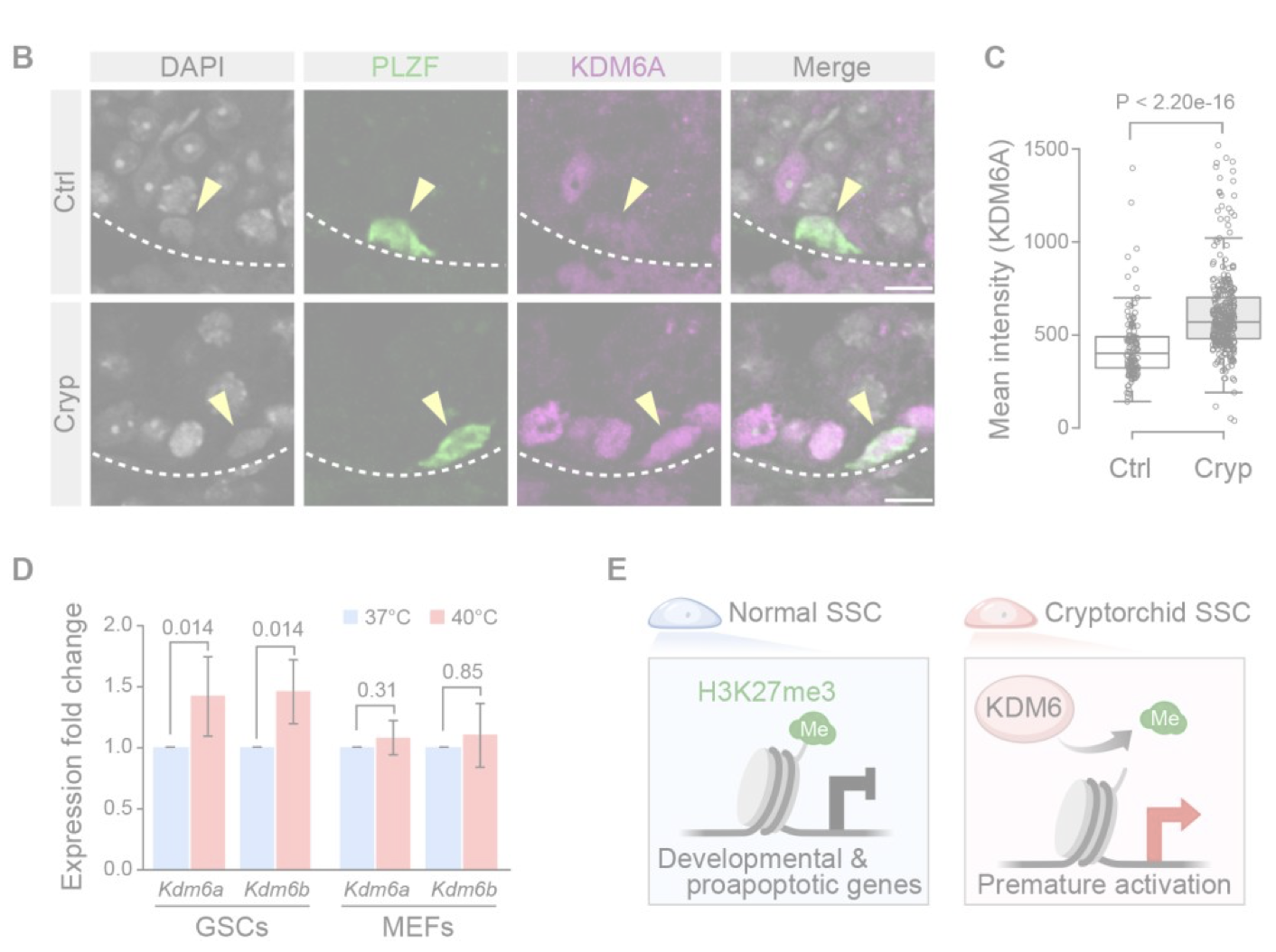
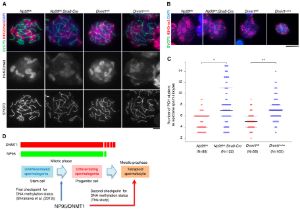
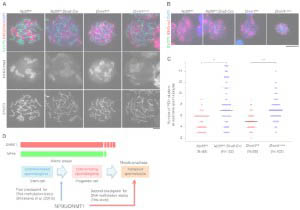
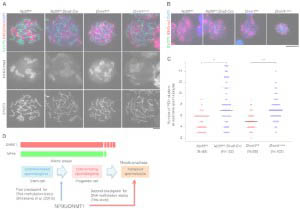
Abnormal H3K27me3 underlies degenerative spermatogonial stem cells in cryptorchid testis.
Kazushige Kuroha, Ivana Dočkal, Uroš Radović, Kuniko Nakajima, Ikue Hoshi, Shion Matsuda, Noriko Kojitani, Kazuyuki Ohbo, Shin-ichi Tomizawa
Gene Expr Patterns (2024) 54, 119383
表紙に選ばれました.
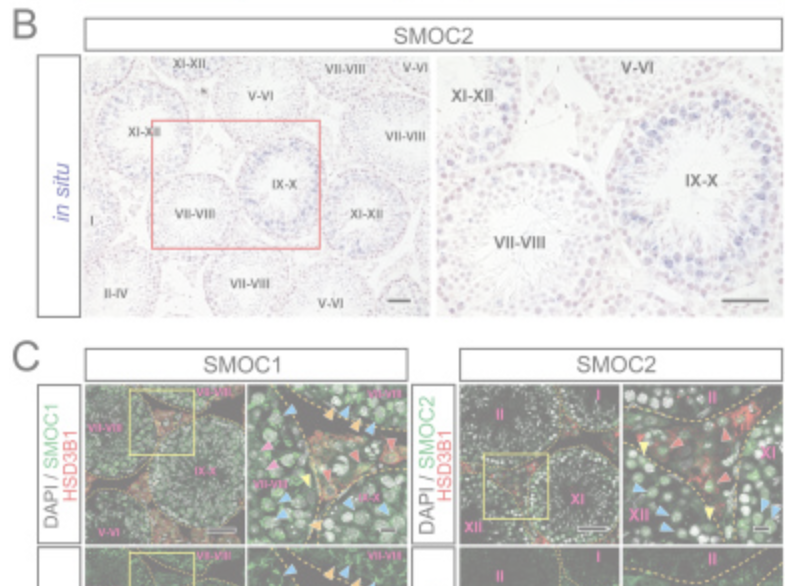
Spatial and temporal expression analysis of BMP signal modifiers, Smoc1 and Smoc2, from postnatal to adult developmental stages in the mouse testis.
M Ono, K Nakajima, S Tomizawa, T Shirakawa, I Okada, H Saitsu, N Matsumoto , K Ohbo
Development (2024) 151(18): dev202834
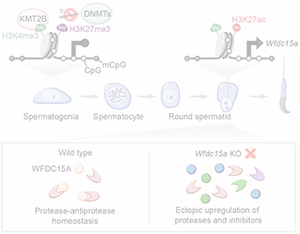
A non-canonical bivalent gene Wfdc15a controls spermatogenic protease and immune homeostasis.
S. Tomizawa, R. Fellows, M. Ono, K.e Kuroha, I. Dockal, Y. Kobayashi, K. Minamizawa, K. Natsume, K. Nakajima, I. Hoshi,S. Matsuda, M. Seki, Y. Suzuki, K. Aoto, H. Saitsu, K. Ohbo
Clinical Epigenetics 13, Article number: 132 (2021)
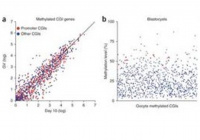
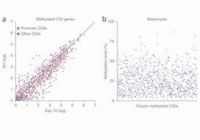
Oxygen concentration affects de novo DNA methylation and transcription in in vitro cultured oocytes.
Florence Naillat, Heba Saadeh, Joanna Nowacka-Woszuk, Lenka Gahurova, Fatima Santos, Shin-ichi Tomizawa & Gavin Kelsey.
Development (2021) 148, dev194605.
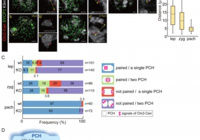
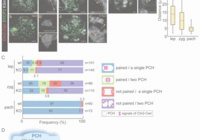
Maintenance DNA methylation in pre-meiotic germ cells regulates meiotic prophase by facilitating homologous chromosome pairing.
Takada Y, Yaman-Deveci R, Shirakawa T, Shari J, Tomizawa S, Miura F, Ito T, Ono M, Nakajima K, Koseki Y, Shiotani F, Ishiguro K, Ohbo K, Koseki H.
Development (2021) 148 (8): dev196212.
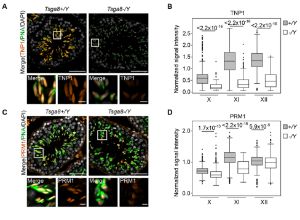
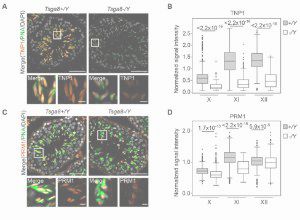
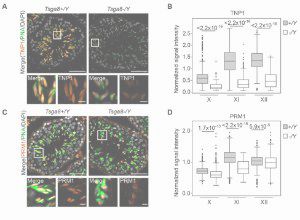
Tsga8 is required for spermatid morphogenesis and male fertility in mice.
Kobayashi Y, Tomizawa S, Ono M, Kuroha K, Minamizawa K, Natsume K, Dizdarević S, Dočkal I, Tanaka H, Kawagoe T, Seki M, Suzuki Y, Ogonuki N, Inoue K, Matoba S, Anastassiadis K, Mizuki N, Ogura A, Ohbo K.
Dis Model Mech (2019) 12 (11): dmm040139.
Lack of whey acidic protein four disulphide core (WFDC) 2 protease inhibitor causes neonatal death from respiratory failure in mice.
Nakajima K, Ono M, Radović U, Dizdarević S, Tomizawa SI, Kuroha K, Nagamatsu G, Hoshi I, Matsunaga R, Shirakawa T, Kurosawa T, Miyazaki Y, Seki M, Suzuki Y, Koseki H, Nakamura M, Suda T, Ohbo K.
Development 145 (2018)
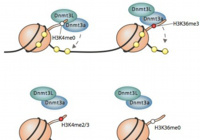
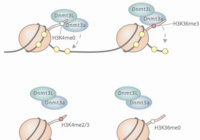
Kmt2b conveys monovalent and bivalent H3K4me3 in spermatogonial stem cells at germline and embryonic promoters.
Tomizawa S, Kobayashi Y, Shirakawa T, Watanabe K, Mizoguchi K, Hoshi I, Nakajima K, Nakabayashi J, Singh S, Dahl A, Alexopoulou D, Seki M, Suzuki Y, Royo H, Peters AHFM, Anastassiadis K, Stewart AF, Ohbo K
Mol Cell Biol. 37(19): e00082-17 (2017)
EPC1/TIP60-Mediated Histone Acetylation Facilitates Spermiogenesis in Mice.
Dong Y, Isono KI, Ohbo K, Endo TA, Ohara O, Maekawa M, Toyama Y, Ito C, Toshimori K, Helin K, Ogonuki N, Inoue K, Ogura A, Yamagata K, Kitabayashi I, Koseki H.
Epigenetics & Chromatin 10: 25-44 (2017)
Transcription and chromatin determinants of de novo DNA methylation timing in oocytes
Gahurova L*, Tomizawa S*, Smallwood SA, Stewart-Morgan KR, Saadeh H, Kim J, Andrews S, Chen T, Kelsey G *Contributed equally
Genome Biology 16: 209-215 (2015)
Deep sequencing and de novo assembly of the mouse oocyte transcriptome define the contribution of transcription to the DNA methylation landscape.
Lenka Veselovska, Sebastien A. Smallwood, ... Shin-ichi Tomizawa4, Simon Andrews and Gavin Kelsey*
BioMol Concepts 6(1): 1–9 2015
Epigenetic regulation in stem cell development, cell fate conversion, and reprogramming.
Kazuyuki Ohbo and Shin-ichi Tomizawa
BMC Genomics 16:624-40 2015
DNA methylation and gene expression dynamics during spermatogonial stem cell differentiation in the early postnatal mouse testis.
Naoki Kubo, ……., Takayuki Shirakawa, Hidetoshi Sone, Yasuyuki Sato, Shin-ichi Tomizawa, Kazuyuki Ohbo
Dev Cell., 34: 1-12 2015
The RNA binding protein Nanos2 organizes a post-transcriptional buffering system to retain primitive state of mouse spermatogonial stem cells.
Zhou, Z., Shirakawa, T., Ohbo, K., Sada, A., Wu, Q., Hasegawa, K., Saba, R. and Saga, Y.
Curr. Pathobiol. Rep., 2:1-9 2014
Stem Cell Epigenetics: Insights from Studies on Embryonic, Induced Pluripotent, and Germline Stem Cells.
Shin-ichi Tomizawa, Takayuki Shirakawa, Kazuyuki Ohbo
Development, 140:3565-3576. 2013
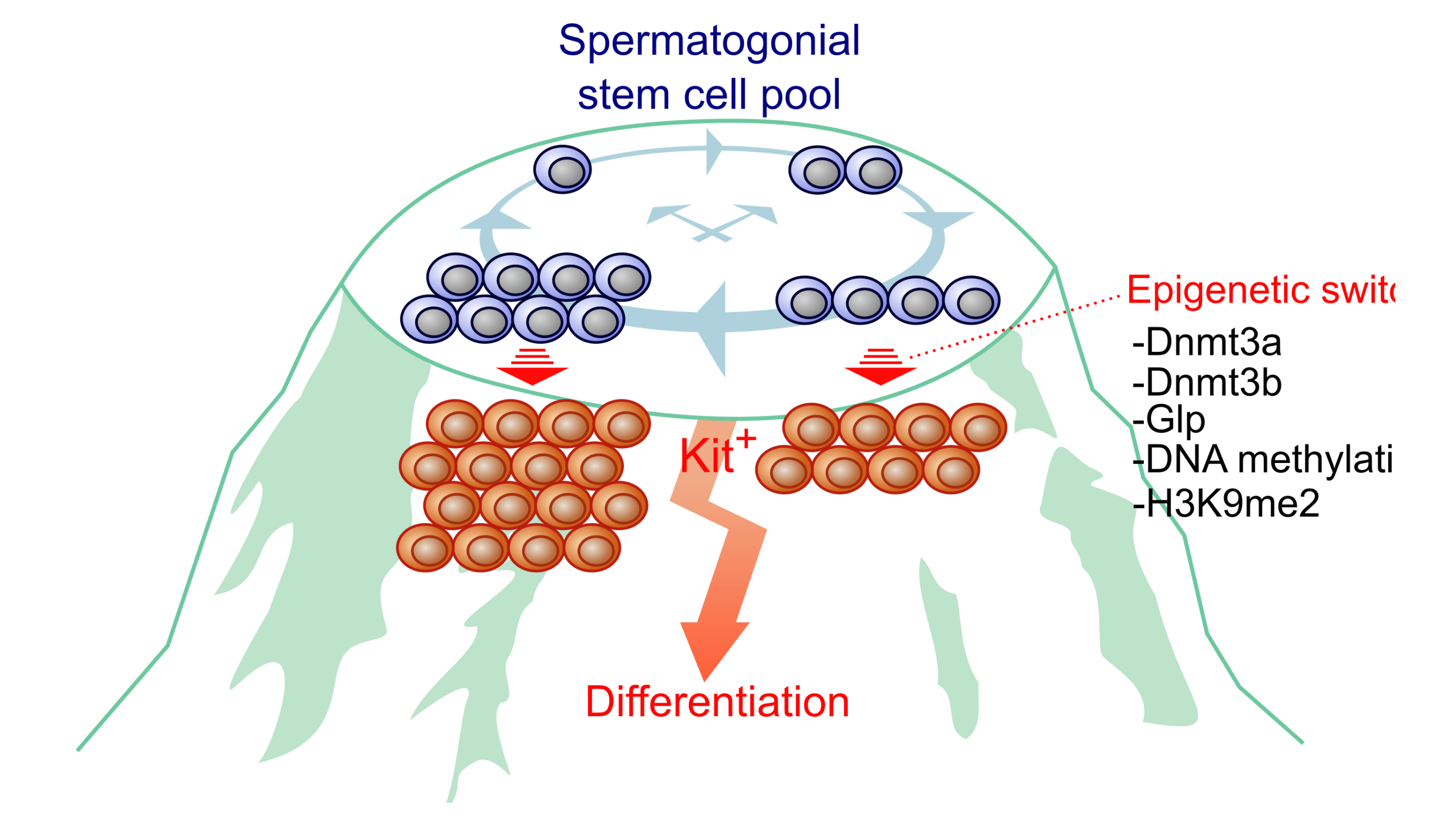
An epigenetic checkpoint controls the transition from a stem cell pool to a progenitor cell state in mouse male germ cells.
Shirakawa T., Yaman-Deveci R., Tomizawa S., Kamizato Y., Nakajima K., at.,al.
The International journal of developmental biology 56 867-875 2012
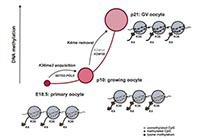
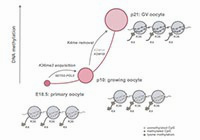
DNA methylation establishment during oocyte growth: mechanisms and significance.
Tomizawa S, Nowacka-Woszuk J, Kelsey G
Nature genetics 43 811-814 2011
Dynamic CpG island methylation landscape in oocytes and preimplantation embryos.
Smallwood SA, Tomizawa S, Krueger F, Ruf N, Carli N, Segonds-Pichon A, Sato S, Hata K, Andrews SR, Kelsey G
Development, 138:4207-4217. 2011
HP1γ links histone methylation marks to meiotic synapsis in mice.
Takada Y., Naruse C., Costa Y., Shirakawa S., Tachibana M., Sharif J., Kezuka-Shiotani F., Kakiuchi D., Masumoto H., Shinkai Y., Ohbo K., Peters A. H.F.M., Turner J. M.A., Asano M. and Koseki K.
Science 332 848-852 2011.
Role for piRNAs and noncoding RNA in de novo DNA methylation of the imprinted mouse Rasgrf1 locus.
Watanabe T, Tomizawa S, Mitsuya K, Totoki Y, Yamamoto Y, Kuramochi-Miyagawa S, Iida N, Hoki Y, Murphy PJ, Toyoda A, Gotoh K, Hiura H, Arima T, Fujiyama A, Sado T, Shibata T, Nakano T, Lin H, Ichiyanagi K, Soloway PD, Sasaki H
Epigenetics & Chromatin 2:5, 2009.
The histone3 lysine 4 methyltransferase, Mll2, is only required briefly in development and spermatogenesis.
Glaser S., Lubitz S., Loveland KL., Ohbo K., Robb L., Schwenk F., Seibler J., Roellig D, Kranz A., Anastassiadis K. and Stewart AF.
Mol Cell Biol. 26:8498-506. 2006.
A CTX family cell adhesion molecule, JAM4, is expressed in stem cell and progenitor cell populations of both male germ cell and hematopoietic cell lineages.
Nagamatsu G, Ohmura M, Mizukami T, Hamaguchi I, Hirabayashi S, Yoshida S, Hata Y, Suda T, and Ohbo K.
Development, 133:1495-1505. 2006.
The first round of mouse spermatogenesis lacks the stem cell stage.
Yoshida S, Sukeno, M, Nakagawa T, Ohbo K, Nagamatsu G, Suda T. and Nabeshima Y-I.




大保 和之


富澤 信一


尾野 道男


黒羽 一誠


中島 久仁子


星 郁栄


林 亜葵


麹谷 典子


南澤 恵佑


松田 志穏


松田 菜那


柏木 悠我
- 博士課程:白川、小林裕貴
- 修士課程:Monika Šafhauzer、Ivana Dockal、Selma Dizdarevic、夏目 幸治、溝口 敬太、Uros Radvic
- 卒研生:前川友紀、渡辺、杉山
- リサクラ:熊谷 凌、藤澤賢太郎

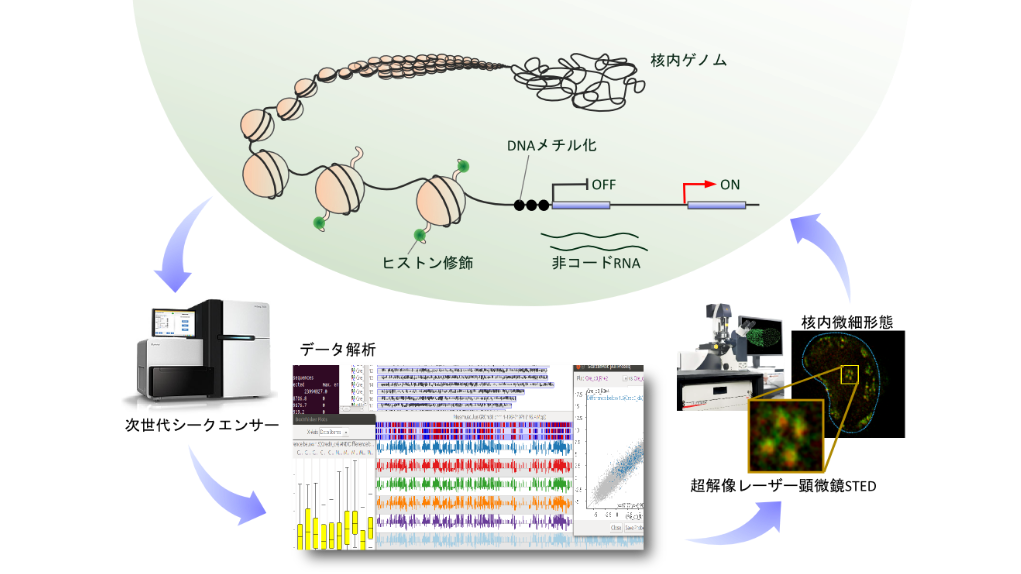
精巣の細胞は体外での幹細胞の長期培養、幹細胞活性を測定するための精細管移植法なども樹立されており、組織学的検索も完成されており、様々な解析手法からアプローチできる細胞系譜です。これらのメリットを生かして研究を行っています。他の教室であまり行われていない手法として、電子顕微鏡、超解像顕微鏡を用いて超解像度の解析も行っています。
- ゲノム修飾(エピジェネティクス)による幹細胞の運命決定制御機構の解析
- 幹細胞の増殖制御とニッチ形成機構の解明
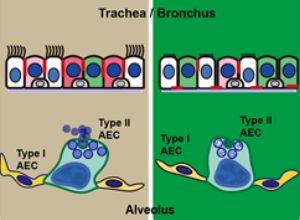
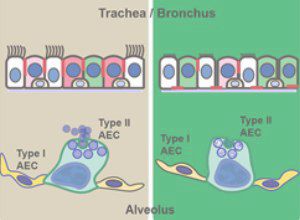
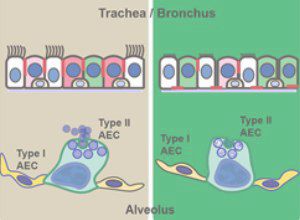
- 肺上皮障害の発症機構の解析
研究室見学、大学院入学など、当研究室に関するお問い合わせは
以下までお気軽にご連絡下さい。
→大学院希望者の方へ
Contact us / お問い合わせ: Tel : 045-787-2567 Fax : 045-787-2568
mail : soshiki1@yokohama-cu.ac.jp
page: http://www-user.yokohama-cu.ac.jp/~finemorp
アウトライン
| 教室名 | 横浜市立大学医学部組織学 |
|---|---|
| 住所 | 横浜市金沢区福浦3−9 |
| 教授 | 大保 和之 |
| 専門 | 解剖学、組織学、エピジェネティクス、生殖細胞、細胞生物学など |
| 技術 | 電子顕微鏡、超解像度顕微鏡、遺伝子解析など |
| 教室員 | 教員4名、職員等4名 |