遺伝学教室の基盤技術
Short-read NGS

ショートリード型次世代シークエンサーを用いた単一遺伝子疾患の遺伝子解析
ショートリード型次世代シークエンスは数百塩基に断片化されたDNAの塩基配列を一度に大量に読む解析手法です。本法は2005年に登場し、単一遺伝子疾患の原因解明に多大な貢献をしていますが、現在では遺伝学研究だけではなく、臨床診断や治療方針の決定を目的とした利用もされており、臨床検査としての利用が始まってきています。当研究室においては2009年にショートリード型次世代シークエンサーの利用を開始し、遺伝子のコード領域をターゲットとした全エクソーム解析を行ってきました。現在ではてんかん、知的障害、多発奇形症候群、神経疾患、その他様々な疾患の全エクソーム解析を行っています。全エクソームシークエンスのデータは1塩基置換や小さいサイズの挿入欠失の検出だけではなく、遺伝子コピー数解析(図1)、連鎖解析なども行うことが可能で、これらの解析から新規の疾患原因遺伝子を多数同定しています。全エクソーム解析によって原因が判明するのは解析症例のうち30-40%と言われていますが、当教室においても同様であり、60-70%の原因不明である症例においては別の解析による原因検索が必要となります。
*バリアント:以前は変異と言われていましたが、現在では病的バリアントまたはバリアントといいます。

体細胞バリアント解析
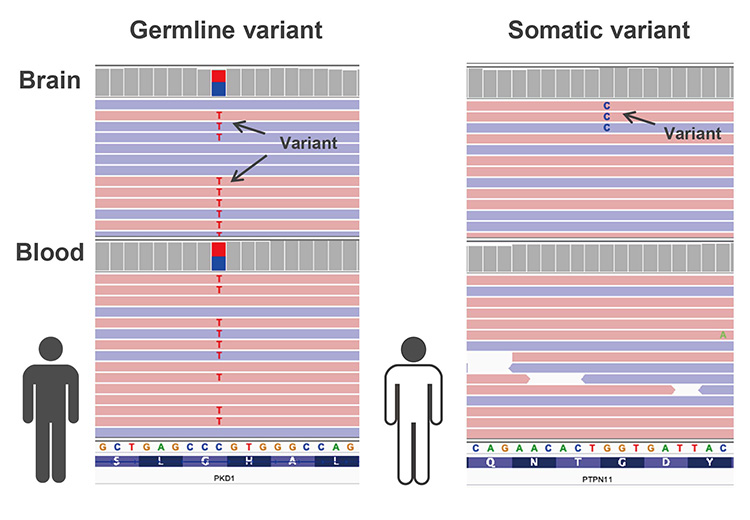
単一遺伝子疾患の遺伝子解析は主に生殖細胞系列のバリアントを対象としています。これらは両親から遺伝したか、受精前の配偶子形成時、あるいは受精卵形成時に起こったバリアント(de novo)であり、患者さんの全身のどの細胞にも存在します(図2)。そのため、精製度が高く、DNAが多く得られて侵襲性の少ない末梢血を用いて解析を行います。しかし、受精後に起こったバリアントである体細胞バリアントによって起こる単一遺伝子疾患も知られています。体細胞バリアントは病変部組織特異的に認めることが多いため、病変部組織からDNAを抽出し、血液由来DNAとの比較によって体細胞バリアントを検出し(図2)、遺伝学的な原因の同定を行います。
Genetic analysis for monogenic diseases using short read next generation sequencer
Short-read next-generation sequencing is an analysis method that is massively parallel sequencing for several hundred base DNA fragments. This method has been commercially available since 2005 and has contributed significantly to human genetics research. Now, it is beginning to be used as a clinical test in Japan, and is being used not only for genetic research, but also for clinical diagnosis and treatment decisions. In our laboratory, we started to use short-read next-generation sequencers in 2009 and have performed whole-exome sequencing (WES) for epilepsy, intellectual disability, multiple congenital anomalies, neurological disorders, and various other diseases. WES data can be used not only to detect single nucleotide substitutions and small insertions and deletions, but also to perform gene copy number analysis (Figure 1) and linkage analysis. From these analyses, we have identified many new disease-causing genes. In our laboratory, the genetic cause of the disease is found by WES in 30-40% of cases, which is similar to other laboratories. In the remaining 60-70% of cases where the cause of the disease is unknown, the other analysis method is required to find the genetic cause.

Somatic variant analysis
The genetic analysis for the monogenic disease usually focuses on germline variants. Germline variants are inherited from either parent or occur at the time of gametogenesis before fertilization or of firtilization (de novo variants). Their variants exist in every cell of the patient’s body (Figure 2). Thus we usually use the DNA derived from peripheral blood, which is high quality and high yield. However, the monogenic disease caused by variants that occur after fertilization, called somatic variants, are also known. Somatic variants often have tissue-specific variants, so DNA is extracted from the lesion tissue and compared with blood-derived DNA to detect tissue-specific variants and identify the causative pathogenic variant (Figure 2).





