遺伝学教室が取り組む技術
Long-read NGS

ロングリードシーケンス解析による未診断症例の原因解明
稀なメンデル遺伝病の半分以上が、今なお未診断とされています。疾患を引き起こす遺伝子変異の種類やサイズは様々で、多様なゲノム変化を網羅的に検出するゲノム解析技術が存在しない事が原因の一つです。
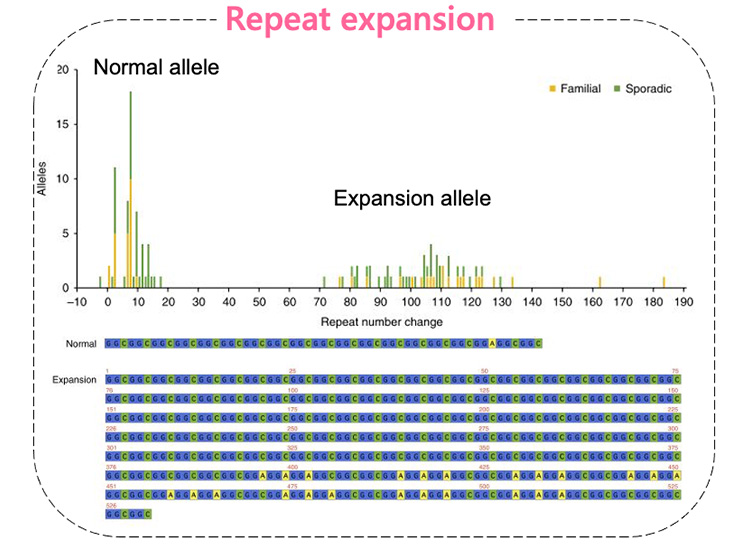
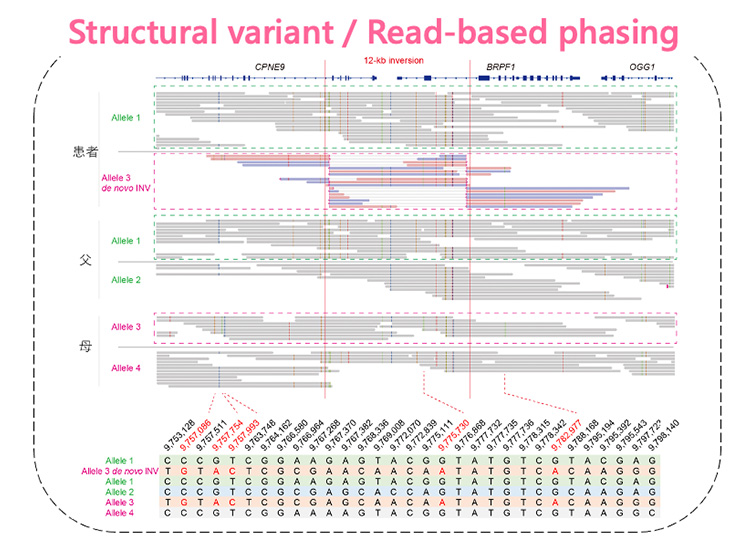
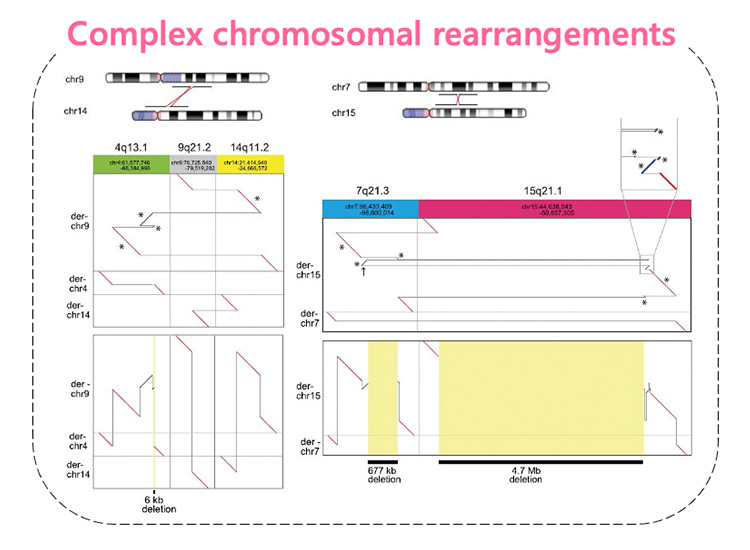
現在、遺伝解析の主流となっている次世代ショートリードシーケンサーは150 bp程度の短いDNA断片を大量かつ同時並行に読むことで疾患原因となる点変異(一塩基置換)を同定するのに優れています。しかし短いリード長のショートリード解析ではゲノム難読領域(同じ塩基単位の繰り返しからなるリピート配列や GCリッチ配列など)の解読や、リード長を超えるサイズのゲノム構造変化(リピート伸長、挿入、欠失、重複、転座、逆位)を十分な感度と精度で検出することが困難です。この点、ロングリードシーケンサーは20,000 bp以上の長いDNA配列を一本のリードで解読することで、既存の解析法で検出が難しい種類・サイズの病的変異(図1、2)や複雑な染色体構造異常の全体像(図3)を明らかにすることが可能です。当教室では、原理の異なる2つのロングリードシーケンサー(PacBio社のSequel, Sequel IIおよびOxford Nanopore社のPromethION, GridION , MinION nanopore sequencer)を用いたゲノム解析系を構築し、未診断症例の原因解明に取り組んでいます。このような未診断症例の新たな遺伝要因を解明する事は遺伝子診断率の向上に直結し、遺伝情報を用いた医療の実現に欠かせない研究課題と考えます。
Elucidation of genetic causes of undiagnosed cases by long-read sequencing
More than half of the rare Mendelian genetic diseases are still undiagnosed. This is partly due to the fact that the types and sizes of disease-causing genetic mutations vary widely, and current genetic/genomic analysis tools cannot reliably detect the entire size range of genomic variation.
Next-generation short-read sequencers, which are currently the mainstream of genetic analysis, are excellent for identifying disease-causing “point” mutations (single nucleotide substitutions) by reading large numbers of short DNA fragments of about 150 bp in parallel. However, short read length (around 150 bp) and biased read coverage (low coverage at repetitive and GC-rich regions) limits their ability to detect certain types of pathogenic variation by NGS.
In this regard, long read sequencers can decode DNA sequences longer than 20,000 bp with a single read, making it possible to reveal new types and sizes of pathological mutations (Figs. 1 and 2) and complex chromosomal structural abnormalities (Fig. 3) that are undetectable by existing analysis methods. We have constructed a genome analysis system using two long read sequencers (Sequel and Sequel II from PacBio and PromethION, GridION, and MinION nanopore sequencers from Oxford Nanopore Technologies) with different principles for sequencing DNA to elucidate the cause of undiagnosed cases. Elucidation of new genetic factors in such undiagnosed cases will directly lead to improvement in the rate of genetic diagnosis, and is an essential research issue for the realization of future precision medicine using genetic information.







